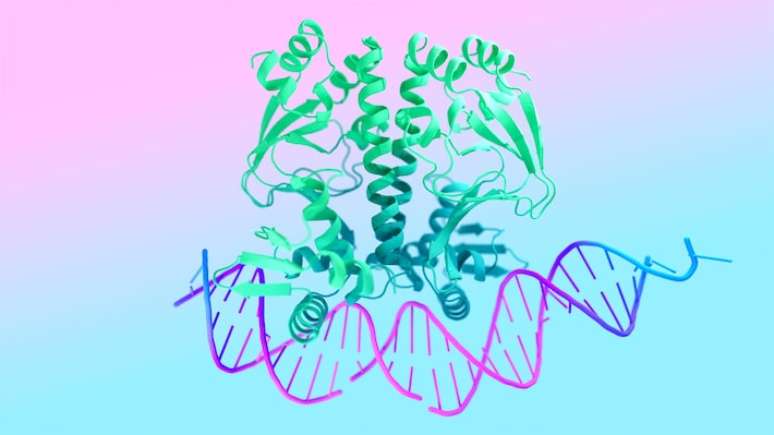
IA do Google revolucionou a ciência das proteínas, mas não 'zerou o jogo'; entenda
Há três anos, o AlphaFold realizou o maior avanço da inteligência artificial na ciência até o momento
Em dezembro de 2020, quando os bloqueios da pandemia impossibilitaram reuniões presenciais, centenas de cientistas da computação se reuniram em frente às suas telas para assistir ao desenrolar de uma nova era da ciência.
Na conferência, realizada a cada dois anos, os cientistas colocaram à prova suas mais recentes ferramentas de dobramento de proteínas. Mas uma solução sempre esteve fora de alcance. Alguns deles haviam passado toda a sua carreira tentando melhorar cada vez mais essas previsões. Essas competições foram marcadas por passos de bebê, e os pesquisadores tinham poucos motivos para pensar que 2020 seria diferente.
Eles estavam errados quanto a isso.
Naquela semana, um recém-chegado à comunidade de ciência de proteínas chamado John Jumper apresentou uma nova ferramenta de inteligência artificial, o AlphaFold2, que surgiu nos escritórios do Google DeepMind, o braço de inteligência artificial (IA) da empresa de tecnologia em Londres. No Zoom, ele apresentou dados que mostravam que os modelos preditivos do AlphaFold2 de estruturas de proteínas em 3D eram mais de 90% precisos - cinco vezes melhores do que os de seu concorrente mais próximo.
Em um instante, o problema de dobragem de proteínas passou de impossível a indolor. O sucesso da inteligência artificial onde a mente humana havia se atrapalhado abalou a comunidade de biólogos. "Fiquei em choque", disse Mohammed AlQuraishi, biólogo de sistemas do Programa de Genômica Matemática da Universidade de Columbia, que participou da reunião. "Muitas pessoas estavam em negação."
Mas, nas observações finais da conferência, seu organizador, John Moult, deixou pouca margem para dúvidas: O AlphaFold2 havia "resolvido em grande parte" o problema de dobramento de proteínas - e mudado a ciência das proteínas para sempre. Sentado em frente a uma estante de livros em seu escritório em casa, vestindo uma gola alta preta e clicando em seus slides no Zoom, Moult falou em um tom animado, mas também ameaçador. "Isso não é um fim, mas um começo", disse ele.
Isso foi há três anos e meio. Finalmente é possível começar a responder a essa questão.
Parte 1
Formulação do problema
Uma longa cadeia de moléculas de aminoácidos não tem função até que se dobre espontaneamente em sua forma inata, que os biólogos chamam de estrutura. A estrutura de uma proteína determina como ela se liga ou interage com outras moléculas e, portanto, define seu papel em uma célula.
"Desde o átomo até os ecossistemas, [a estrutura da proteína] é uma espécie de língua franca", disse AlQuraishi. "É onde tudo acontece."
Se as proteínas não realizassem esse processo de dobragem extremamente bem, uma cascata de desastres se espalharia pelo corpo. Uma proteína dobrada ou desdobrada incorretamente pode levar à toxicidade e à morte celular. Muitas doenças e distúrbios, como a anemia falciforme, são causados por proteínas mal dobradas. E as proteínas mal dobradas podem se agregar em aglomerados que são marcas registradas de doenças neurodegenerativas como Alzheimer e Parkinson.
No entanto, ninguém sabe ao certo como ocorre o dobramento das proteínas. Como as informações da sequência nessas cadeias moleculares simples codificam a forma complexa de uma proteína? Essa é a "pergunta mais profunda que podemos fazer", disse George Rose, professor emérito de biofísica da Universidade Johns Hopkins.
Depois que sua cadeia polipeptídica é montada, uma proteína pode se dobrar em sua estrutura em um milésimo de segundo. Com o passar do tempo, o problema do dobramento de proteínas se dividiu em novos tipos de problemas. Agora, três questões principais são colocadas: A estrutura de uma proteína pode ser prevista a partir de sua sequência de aminoácidos? Qual é o código de dobramento? E qual é o mecanismo de dobragem?
À medida que surgiam mais estruturas, a comunidade da ciência das proteínas precisava de uma maneira de organizá-las e compartilhá-las. Em 1971, o Protein Data Bank foi fundado como um arquivo de estruturas de proteínas. Disponível gratuitamente, o banco de dados tornou-se uma ferramenta confiável para qualquer pessoa que precisasse conhecer a estrutura de uma proteína para investigar uma questão biológica.
Quando o Protein Data Bank foi inaugurado, ele continha as estruturas de sete proteínas. Quando o Google DeepMind o utilizou para treinar o AlphaFold2, quase 50 anos depois, ele continha mais de 140.000 - cada uma delas laboriosamente decodificada pelos cientistas conhecidos como biólogos estruturais.
Conferência tem início
No início da década de 1990, John Moult, pesquisador da Universidade de Maryland, e seu colega Krzysztof Fidelis tiveram uma ideia para trazer disciplina ao caos do campo. Eles criaram um experimento científico comunitário que chamaram de Avaliação Crítica da Previsão de Estrutura, ou CASP, na sigla em inglês.
A ideia era simples. Como organizadores do CASP, Moult e Fidelis publicariam uma lista de sequências de aminoácidos para proteínas cujas estruturas tivessem sido resolvidas recentemente e fornecidas a eles por experimentalistas, mas cujos resultados ainda não tivessem sido publicados. Em seguida, grupos de computação de todo o mundo fariam o possível para prever a estrutura da proteína usando o método que desejassem. Um grupo independente de cientistas avaliaria os modelos comparando suas respostas com as estruturas confirmadas experimentalmente.
Em 1996, após o término do segundo CASP, um jovem chamado David Baker pediu a David Jones, professor de bioinformática da University College London, para dividir um táxi até o aeroporto. Baker havia assistido à palestra de Jones e estava trabalhando em seu próprio modelo computacional. Ele não estava pronto para esse CASP, mas queria conversar sobre ele. Jones ouviu suas ideias no táxi e não esperava vê-lo novamente.
Na competição seguinte, em 1998, Baker abriu as portas com seu algoritmo Rosetta. Ele se tornou "o homem a ser batido", disse Jones.
Algoritmos como o Rosetta modelaram as interações entre os átomos das moléculas de aminoácidos para prever como elas se dobrariam. Eles "mostraram que realmente era possível prever a estrutura das proteínas", disse Baker. "Mas não era bom ou preciso o suficiente para ser útil."
Os biólogos computacionais aprimoraram as ferramentas estatísticas. Em 2016, a precisão da previsão de contatos subiu para 47%. Dois anos depois, chegou a 70%. O algoritmo de Baker se baseou nesse sucesso: Em 2014, o Rosetta produziu duas estruturas de proteínas tão precisas que um avaliador do CASP achou que Baker poderia ter resolvido o problema de dobramento de proteínas.
No entanto, isso levou o campo apenas até certo ponto. A coevolução exigia uma abundância de proteínas semelhantes para comparação, e os experimentalistas não estavam resolvendo estruturas de proteínas com rapidez suficiente para suprir as necessidades dos computacionalistas.
"Eu esperava que [o AlphaFold2] fosse cair", disse Janet Thornton, biofísica da Universidade de Oxford. "Mas, na verdade, minha impressão é que ele tem tido um sucesso extraordinário."
Começou a ficar claro que, em vez de ser uma ameaça, o AlphaFold2 poderia ser um catalisador para acelerar a pesquisa. Em vez de tirar o emprego dos biólogos estruturais, ela lhes proporcionou uma nova ferramenta para fazer melhor seu trabalho.
No entanto, isso não resultou imediatamente em todos os tipos de novos medicamentos, como algumas pessoas previram, e os pesquisadores logo perceberam que a ferramenta tem suas limitações. As previsões do AlphaFold2 não são perfeitas. Elas exigem validação experimental, disse Perrakis. Mas "você pode passar muito mais rápido para o estudo real das estruturas". Agora, quando seus alunos iniciam um novo projeto, eles primeiro usam o AlphaFold2 para prever a estrutura de uma determinada proteína. Em seguida, realizam experimentos para validá-la.
Perrakis suspeita que ele e outros pesquisadores continuarão a usar a cristalografia de raios X até certo ponto. Mas, para desenvolver estruturas iniciais de proteínas, muitos estão começando a combinar previsões de aprendizagem profunda com técnicas avançadas de microscópio eletrônico, como o cryo-EM, que envolve o congelamento de amostras biológicas e seu bombardeio com elétrons. Assim, eles podem chegar às questões interessantes sobre o que suas proteínas fazem. O AlphaFold2 "turbinou" o crio-EM, disse AlQuraishi.
Essa mudança já começou. Em junho de 2022, uma edição especial da revista Science revelou a estrutura quase atômica de um complexo de poros nucleares humano. Essa estrutura maciça e complicada - formada por 30 proteínas diferentes - foi um dilema biológico durante décadas. Os cientistas usaram as previsões do AlphaFold2 para preencher as lacunas nas estruturas das proteínas que não foram resolvidas pelo cryo-EM.
Ver esse artigo, no qual outros cientistas usaram o AlphaFold2 para fazer uma descoberta biológica, foi o "momento em que eu soube que [o AlphaFold] era realmente importante", disse Jumper.
A próxima fronteira
Quase imediatamente depois de ver a palestra de Jumper na conferência CASP de 2020, Baker voltou a trabalhar em seu algoritmo Rosetta. O Google ainda não havia compartilhado o código-fonte subjacente do AlphaFold2. Mesmo assim, "começamos a brincar com algumas das ideias que eles apresentaram", disse Baker. No mesmo dia em que o Google DeepMind publicou o AlphaFold2 na Nature, ele e sua equipe anunciaram o RoseTTAFold, um rival altamente preciso do AlphaFold. O RoseTTAFold também usa deep learning para prever estruturas de proteínas, mas tem uma arquitetura subjacente muito diferente do AlphaFold2.
A RoseTTAFold não estava sozinha. Outros concorrentes da AlphaFold, incluindo a Meta, criaram seus próprios algoritmos para abordar a previsão da estrutura de proteínas ou problemas relacionados. Alguns, incluindo a startup de biotecnologia Atomic AI, de Townshend, foram além das proteínas e usaram o aprendizado profundo para entender as estruturas de RNA. Entretanto, no âmbito das previsões de estrutura única, ninguém conseguiu igualar a precisão do AlphaFold até o momento, disse Thornton. "Tenho certeza de que conseguirão, mas acho que será muito difícil conseguir outro momento (...) do AlphaFold como esse."
Os biólogos estão apenas começando a testar essas atualizações. Até agora, o AlphaFold3 é muito mais preciso do que o RoseTTAFold All-Atom, disse AlQuraishi - mas não é um salto tão grande quanto um "momento AlphaFold2?. Para algumas macromoléculas, como as estruturas de RNA, sua precisão permanece abaixo da de outros sistemas e experimentos baseados em física.
Mesmo assim, os novos algoritmos são um passo na direção certa. As interações entre proteínas e outras moléculas são essenciais para seu funcionamento nas células. Para desenvolver medicamentos que possam se acoplar às proteínas e alterar sua atividade conforme desejado, os pesquisadores precisam entender como são esses complexos. No entanto, é improvável que qualquer um dos algoritmos leve a novos medicamentos em breve, disse Adams. "Ambos os métodos ainda são limitados em sua precisão, [mas] ambos são melhorias significativas em relação ao que era possível."
Há uma outra grande mudança no novo produto da DeepMind. O código subjacente do AlphaFold2 era de código aberto para que outros pesquisadores pudessem estudar o algoritmo e refazê-lo para seus próprios projetos. No entanto, em vez de compartilhar o código-fonte do AlphaFold3, o Google optou, até o momento, por protegê-lo como um segredo comercial. "Pelo menos por enquanto, ninguém pode executá-lo e usá-lo como fizeram com o [AlphaFold2]", disse AlQuraishi.
Mesmo antes do lançamento do AlphaFold3, os pesquisadores estavam testando o AlphaFold2 para ver se ele poderia fornecer informações úteis sobre proteínas em diferentes conformações. Brenda Rubenstein, professora associada de química e física da Brown University, estava interessada em quinases, um tipo de proteína que ativa outras proteínas. Especificamente, ela queria entender o mecanismo de uma quinase que causa câncer para que pudesse desenvolver medicamentos mais precisos contra ela. Seu laboratório modelou a estrutura da quinase usando uma abordagem baseada na física, que mapeia as coordenadas 3D dos átomos usando as leis de Newton. Isso levou dois anos e meio.
"Há cerca de um ano, dissemos: podemos fazer isso mais rápido?" disse Rubenstein. Eles tentaram usar o AlphaFold2 de uma forma inovadora. Ao fornecer dados sobre proteínas relacionadas ao algoritmo, ela descobriu que ele podia prever sua quinase em diferentes conformações com mais de 80% de precisão.
A visão de alguém de fora
Quando o CASP 2020 chegou, muitos pesquisadores esperavam que um avanço na previsão de estruturas viesse por meio da inteligência artificial. "Tudo estava caminhando nessa direção", disse Townshend. Mas eles não esperavam que isso viesse de uma empresa de tecnologia multibilionária, e não esperavam que fosse tão cedo. Alguns disseram que o AlphaFold2 não era uma façanha da nova ciência, mas sim uma engenharia inteligente. Alguns ficaram surpresos com o fato de os algoritmos de David Baker não terem levado o troféu. Outros ficaram menos surpresos por causa dos recursos inigualáveis do Google DeepMind.
Cerca de 100 laboratórios participam do CASP todos os anos e, embora tenham começado a adotar tecnologias de IA, eles "provavelmente não tinham a experiência que o DeepMind tinha, nem o poder de computação", disse Thornton. O DeepMind "tinha acesso a um poder de computação basicamente ilimitado".
Ela também especulou que a falta de conhecimento especializado do Google em ciência de proteínas pode tê-los liberado de forma criativa. "Eles tinham um único objetivo", disse Thornton, e se concentraram na criação de uma excelente rede neural. Os biólogos de proteínas tinham bagagem. Enquanto trabalhavam em suas ferramentas de IA, eles queriam capturar a física molecular de nível atômico e a química envolvida no dobramento de proteínas. O DeepMind tinha uma abordagem diferente: transformaremos os dados de sequência em uma estrutura 3D, e não importa como chegaremos lá.
O novo mundo
Setenta anos atrás, acreditava-se que as proteínas eram uma substância gelatinosa. O campo da biologia de proteínas é "mais empolgante agora do que era antes do AlphaFold", disse Perrakis. A empolgação vem da promessa de reviver a descoberta de medicamentos com base na estrutura, da aceleração na criação de hipóteses e da esperança de compreender as interações complexas que ocorrem nas células.
"É como a revolução genômica", disse AlQuraishi. Há tantos dados, e os biólogos, seja em seus laboratórios úmidos ou na frente de seus computadores, estão apenas começando a descobrir o que fazer com tudo isso.
Mas, assim como outras descobertas de inteligência artificial que estão surgindo em todo o mundo, essa pode ter um teto.
O sucesso do AlphaFold2 se baseou na disponibilidade de dados de treinamento - centenas de milhares de estruturas de proteínas meticulosamente determinadas pelas mãos de experimentadores pacientes. Embora o AlphaFold3 e os algoritmos relacionados tenham demonstrado algum sucesso na determinação das estruturas de compostos moleculares, sua precisão está aquém da de seus predecessores de proteína única. Isso se deve, em parte, ao fato de haver muito menos dados de treinamento disponíveis.
O problema de dobragem de proteínas foi "quase um exemplo perfeito para uma solução de IA", disse Thornton, porque o algoritmo poderia ser treinado em centenas de milhares de estruturas de proteínas coletadas de maneira uniforme. No entanto, o Protein Data Bank pode ser um exemplo incomum de compartilhamento organizado de dados em biologia. Sem dados de alta qualidade para treinar algoritmos, eles não farão previsões precisas.
"Tivemos sorte", disse Jumper. "Encontramos o problema no momento em que ele estava pronto para ser resolvido."
Ninguém sabe se o sucesso do deep learning na solução do problema de dobramento de proteínas será levado a outros campos da ciência ou mesmo a outras áreas da biologia. Mas alguns, como AlQuraishi, estão otimistas. "O dobramento de proteínas é, na verdade, apenas a ponta do iceberg", disse ele. Os químicos, por exemplo, precisam realizar cálculos caros do ponto de vista computacional. Com a aprendizagem profunda, esses cálculos já estão sendo feitos até um milhão de vezes mais rápido do que antes, disse AlQuraishi.
A inteligência artificial pode claramente avançar em tipos específicos de questões científicas. Mas ela pode levar os cientistas apenas até certo ponto no avanço do conhecimento. "Historicamente, a ciência tem se dedicado a compreender a natureza", disse AlQuraishi - os processos que fundamentam a vida e o universo. Se a ciência avançar com ferramentas de aprendizagem profunda que revelam soluções e nenhum processo, será que isso é realmente ciência?
Se muitos pesquisadores decidirem desistir de compreender os processos da natureza, então a inteligência artificial não terá mudado apenas a ciência - ela terá mudado os cientistas também.
A próxima reunião do CASP será realizada em dezembro de 2024 nas águas do Mar do Caribe. Os ventos são cordiais, como provavelmente será a conversa. O barulho já diminuiu há muito tempo - pelo menos em voz alta. Não se sabe como será a competição deste ano. Mas se os últimos CASPs são alguma indicação, Moult sabe que deve esperar apenas uma coisa: "surpresas".
História original republicada com permissão da Quanta Magazine, uma publicação editorialmente independente apoiada pela Simons Foundation. Leia o conteúdo original em How AI Revolutionized Protein Science, but Didn't End It.
Este conteúdo foi traduzido com o auxílio de ferramentas de Inteligência Artificial e revisado por nossa equipe editorial. Saiba mais em nossa Política de IA.


-s101mh9gjfc1.jpg)